Multiplex ligatie-afhankelijke probeamplificatie (MLPA) is een moleculaire techniek die in 2002 door MRC-Holland is ontwikkeld. In een notendop is MLPA een gevoelige techniek waarmee snel en efficiënt nucleïnezuursequenties kunnen worden gekwantificeerd. De techniek wordt wereldwijd in veel laboratoria toegepast en kan worden gebruikt om veranderingen in het kopienummer (zoals deleties of duplicaties) van een gen op te sporen, de methyleringsstatus van DNA te bepalen, single nucleotide polymorfismen (SNPs) en puntmutaties op te sporen, en mRNA te kwantificeren. Daarom wordt het gebruikt in veel onderzoek en diagnostische gebieden, zoals cytogenetica, kankeronderzoek, en menselijke genetica, onder anderen.
Hoe werkt het?
MLPA bestaat uit de volgende stappen (figuur 1):
- Denaturatie
- Hybridisatie
- Ligatie
- Amplificatie (door PCR)
- Fragmentscheiding en data-analyse
Figuur 1 – Visualisatie van de MLPA-techniek (aangepast van Schouten, Jan P., et al.1). We zien ook een typisch electropherogram verkregen door MLPA analyse dat een deletie van exon 46 toont (rode pijl). (Elektroferogram aangepast uit https://commons.wikimedia.org/wiki/File:MLPA_in_GeneMarker.jpg)
1-Denaturatie en 2 – Hybridisatie
Denaturatie houdt in scheiding van de geannealde DNA-strengen, zodat dubbelstrengs DNA enkelstrengs wordt.
Hybridisatie houdt in hybridisatie van het DNA-monster aan specifieke probes. Omdat het een multiplex-techniek is, kunt u elk monster met wel 60 probes tegelijk analyseren, zodat verschillende plaatsen worden bestreken!
Deze probes hebben een primer-sequentie die zich in het amplificatieproces aan de PCR-primer bindt. Alle verschillende probes hebben dezelfde primer-bindende sequentie. Daarnaast hebben de probes ook een hybridisatiesequentie die complementair is aan de doelplaats, waardoor de probe zich aan het DNA kan binden. Beide probes zullen hybridiseren op aangrenzende plaatsen op de DNA-streng.
Een van de probes van het paar bevat een stuffer-sequentie, die voor elke doelsite verschillend van lengte is. De lengte van de stuffer-sequentie verandert tussen de verschillende probes, waardoor multiplexing mogelijk wordt. U kunt dus verwachten dat elk amplificatieproduct een unieke lengte heeft!
3-Ligatie
De ligatiestap bindt de twee probes aan elkaar. In deze stap wordt een specifiek enzym, DNA-ligase, gebruikt. Het bindt de probes die reeds gehybridiseerd zijn op aangrenzende plaatsen van de DNA-streng op de doellocatie. Het ligase dat in MLPA-protocollen wordt gebruikt is ligase-65, een NAD-afhankelijk ligase-enzym, dat ook in andere toepassingen nuttig kan zijn.
Nu rijst de vraag: als het ons doel is beide probes te ligeren, waarom zijn het dan om te beginnen afzonderlijke moleculen? Wel, beide probes bevatten de bindingsplaatsen voor PCR-primers. Dit betekent dat, als we de probes als één enkel molecuul zouden gebruiken, we een amplificatieproduct zouden verkrijgen, zelfs zonder de DNA-doelplaats, waardoor we een niet-specifieke amplificatie zouden krijgen. Het enzym ligase is uiterst specifiek: indien er mismatches zijn tussen de probe en de doelplaats, zal het ligase niet in staat zijn de probes te binden en zal er geen amplificatie optreden. Bijgevolg kunnen met MLPA specifieke puntmutaties worden opgespoord en kan zelfs onderscheid worden gemaakt tussen pseudogenen en het echte doelgen.
4-Omplificatie
De volgende stap is de amplificatie, die in wezen een polymerase-kettingreactie (PCR) is (tabel 1). Voor de PCR-stap worden een polymerase, dNTPs, en een voorwaartse en een tegenwaartse primer toegevoegd. Aangezien alle probes dezelfde PCR-primersequentie hebben, behoeft slechts één paar universele primers te worden toegevoegd om al onze doelwitten te bestuderen. De voorwaartse primer is fluorescent gelabeld, waardoor visualisatie en kwantificering tijdens de analyse mogelijk zijn.
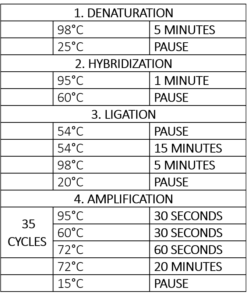
Tabel 1 – Thermocycler-programma voor de MLPA-reactie
5-Fragmentafscheiding en gegevensanalyse
Na de amplificatie worden de fragmenten gescheiden door capillaire elektroforese. Capillaire elektroforese scheidt de fragmenten op basis van hun lengte, en toont de fragmenten van verschillende lengte als piekpatronen, een zogenaamd elektroferogram (figuur 1). Elk amplicon heeft een andere bekende grootte, als gevolg van de stuffer-sequentie op elke specifieke probe, en daarom kan elk amplicon tijdens de gegevensanalyse worden gekwantificeerd.
De gegevens die door capillaire elektroforese worden verkregen, vormen de input voor de analyse. MRC Holland levert gratis software voor data-analyse – Coffalyser.
Door elk monster te vergelijken met een set referentiemonsters, kunnen we een probe ratio verkrijgen. Deze probe ratio vertelt ons hoeveel kopieën een gen heeft. Aangezien de meeste menselijke genen diploïd zijn, zal, als het monster twee kopieën heeft, de ratio 1,0 zijn; d.w.z. dat de sondes van het monster hetzelfde aantal genen hebben verkregen als het referentiemonster.
Als de ratio echter 0,5 is, was er slechts één kopie van het gen in het individu, wat waarschijnlijk een heterozygote deletie van het doelgen betekent. Als de ratio daarentegen 1.5 is, is er waarschijnlijk sprake van een heterozygote duplicatie van een gen.
MRC-Holland biedt veel verschillende kits die mogelijk de oplossing voor uw problemen bieden. Als u echter iets obscuurders wilt vinden, of iets wilt bestuderen dat niet in een kit zit, kunt u uw eigen probes ontwerpen. Ik raad u aan het protocol voor het ontwerpen van synthetische probes aandachtig te lezen.
Voordelen van MLPA
- MLPA is een zeer gevoelige, robuuste en high throughput techniek.
- Het kan onderscheid maken tussen puntmutaties, maar ook tussen duplicatie/deletie van genen. Daarom heeft zij een groot voordeel boven andere technieken, zoals sequencing, die alleen puntmutaties kunnen vinden. Bovendien kan MLPA, in tegenstelling tot FISH, kleine genmutaties opsporen.
- Resultaten zijn binnen 24 uur beschikbaar en omdat het een multiplexreactie is, kan snel en efficiënt informatie worden verzameld.
- Kleine wijzigingen in het MLPA-protocol kunnen een verscheidenheid van toepassingen mogelijk maken. Door bijvoorbeeld een extra digestiestap toe te voegen, kan MLPA ook worden gebruikt om methyleringspatronen in DNA op te sporen (Methyleringsspecifieke-MLPA (MS-MLPA)).
Beperkingen van MLPA
- MLPA is uiterst gevoelig voor onzuiverheden. Daarom is uiterste voorzichtigheid geboden bij het bereiden van monsters en het uitvoeren van de techniek.
- Er kan een afname van het signaal van een probe optreden als gevolg van een zeldzaam polymorfisme of mutatie, en dan kan het nodig zijn dit met andere technieken te testen.
MLPA is een geweldige techniek die voor uiteenlopende toepassingen kan worden gebruikt en snel en efficiënt resultaten oplevert. Hij is echter niet zonder haperingen. Zoals we allemaal weten, heeft elke techniek bepaalde nadelen, en elke toepassing moet grondig worden bestudeerd, om er zeker van te zijn dat we de meest efficiënte en gevoelige techniek gebruiken.
Heb je ooit MLPA gebruikt? Wat zijn uw gedachten over deze techniek?