La Multiplex Ligation-dependent Probe Amplification (MLPA) est une technique moléculaire développée par MRC-Holland en 2002. En quelques mots, la MLPA est une technique sensible qui permet de quantifier des séquences d’acides nucléiques, rapidement et efficacement. Elle est utilisée dans de nombreux laboratoires du monde entier et peut être appliquée pour détecter les modifications du nombre de copies (délétions ou duplications) d’un gène, identifier le statut de méthylation de l’ADN, détecter les polymorphismes de nucléotides simples (SNP) et les mutations ponctuelles, et quantifier l’ARNm. Par conséquent, il est utilisé dans de nombreux domaines de recherche et de diagnostic, tels que la cytogénétique, la recherche sur le cancer et la génétique humaine, entre autres.
Comment fonctionne-t-il ?
L’AMPL comprend les étapes suivantes (figure 1) :
- Dénaturation
- Hybridation
- Ligation
- Amplification (par PCR)
- Séparation des fragments et analyse des données
Figure 1 – Visualisation de la technique MLPA (adaptée de Schouten, Jan P., et al.1). Nous pouvons également observer un électrophérogramme typique obtenu par analyse MLPA montrant une délétion de l’exon 46 (flèche rouge). (Electrophérogramme adapté de https://commons.wikimedia.org/wiki/File:MLPA_in_GeneMarker.jpg)
1-Dénaturation et 2 – Hybridation
La dénaturation consiste à séparer les brins d’ADN recuits, de sorte que l’ADN double brin devient simple brin.
L’hybridation consiste à hybrider l’échantillon d’ADN à des sondes spécifiques. Comme il s’agit d’une technique multiplex, vous pouvez analyser chaque échantillon par jusqu’à 60 sondes simultanément, ciblant ainsi différents sites !
Ces sondes ont une séquence d’amorce qui se lie à l’amorce PCR dans le processus d’amplification. Toutes les différentes sondes auront la même séquence de liaison d’amorce. De plus, les sondes ont également une séquence d’hybridation complémentaire au site cible qui permettra à la sonde de se lier à l’ADN. Les deux sondes vont s’hybrider sur des sites adjacents du brin d’ADN.
L’une des sondes de la paire contient une séquence stuffer, dont la longueur est différente pour chaque site cible. La longueur de la séquence stuffer change entre les différentes sondes, ce qui permet le multiplexage. Ainsi, vous pouvez vous attendre à ce que chaque produit d’amplification ait une longueur unique !
3-Ligation
L’étape de ligature va lier les deux sondes ensemble. Dans cette étape, une enzyme spécifique appelée ADN ligase est utilisée. Elle lie les sondes qui sont déjà hybridées sur des sites adjacents du brin d’ADN au site cible. La ligase utilisée dans les protocoles MLPA est la ligase-65, une enzyme ligase dépendante du NAD, qui peut également être utile dans d’autres applications.
Maintenant, la question se pose : si notre objectif est de ligaturer les deux sondes, pourquoi sont-elles des molécules séparées au départ ? Eh bien, les deux sondes contiennent les sites de liaison pour les amorces PCR. Cela signifie que si nous devions utiliser les sondes comme une seule molécule, nous obtiendrions un produit d’amplification, même sans le site cible de l’ADN, ce qui nous donnerait une amplification non spécifique. L’enzyme ligase est extrêmement spécifique : s’il y a des mésappariements entre la sonde et le site cible, la ligase ne pourra pas lier les sondes et aucune amplification ne se produira. Par conséquent, la MLPA détecte des mutations ponctuelles spécifiques, et fait même la distinction entre les pseudogènes et le véritable gène cible.
4-Amplification
L’étape suivante est l’amplification, qui est essentiellement une réaction en chaîne par polymérase (PCR) (tableau 1). Pour l’étape de la PCR, on ajoute une polymérase, des dNTP et une amorce directe et inverse. Comme toutes les sondes ont la même séquence d’amorces PCR, il ne sera nécessaire d’ajouter qu’une seule paire d’amorces universelles pour étudier toutes nos cibles. L’amorce directe est marquée par fluorescence, ce qui permet la visualisation et la quantification pendant l’analyse.
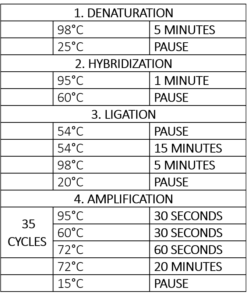
Tableau 1 – Programme du thermocycleur pour la réaction MLPA
5-Séparation des fragments et analyse des données
Après l’amplification, les fragments sont séparés par électrophorèse capillaire. L’électrophorèse capillaire sépare les fragments en fonction de leur longueur, et montre les fragments de différentes longueurs sous forme de pic, appelé électrophérogramme (figure 1). Chaque amplicon a une taille différente connue, en raison de la séquence de bourrage sur chaque sonde spécifique, et donc chaque amplicon peut être quantifié pendant l’analyse des données.
Les données obtenues par électrophorèse capillaire seront l’entrée pour l’analyse. MRC- Holland fournit un logiciel gratuit pour l’analyse des données – Coffalyser.
En comparant chaque échantillon à un ensemble d’échantillons de référence, nous pouvons obtenir un ratio de sonde. Ce ratio de sondes nous informera du nombre de copies d’un gène. Comme la plupart des gènes humains sont diploïdes, si l’échantillon présente deux copies, le ratio sera de 1,0 ; c’est-à-dire que les sondes de l’échantillon ont obtenu le même nombre de gènes que l’échantillon de référence.
En revanche, si le ratio est de 0,5, il n’y avait qu’une seule copie du gène dans l’individu, ce qui signifie probablement une délétion hétérozygote du gène cible. Si, en revanche, le ratio est de 1,5, il y a, probablement, une duplication hétérozygote d’un gène.
MRC-Holland propose de nombreux kits différents qui peuvent avoir la solution à vos problèmes. Cependant, si vous essayez de trouver quelque chose d’un peu plus obscur, ou d’étudier quelque chose qui n’est dans aucun kit, vous pouvez concevoir vos propres sondes. Je vous conseille de lire attentivement le protocole de conception de sondes synthétiques.
Avantages de la MLPA
- La MLPA est une technique très sensible, robuste et à haut débit.
- Elle peut discerner les mutations ponctuelles, ainsi que la duplication/délétion des gènes. Elle présente donc un grand avantage par rapport à d’autres techniques, comme le séquençage, qui ne peuvent trouver que des mutations ponctuelles. De plus, contrairement à la FISH, la MLPA peut détecter de petites altérations génétiques.
- Les résultats sont disponibles dans les 24 heures et, comme il s’agit d’une réaction multiplex, elle permet une collecte rapide et efficace des informations.
- De petites modifications du protocole de la MLPA peuvent permettre une variété d’applications. Par exemple, en ajoutant une étape de digestion supplémentaire, la MLPA peut également être utilisée pour détecter des modèles de méthylation dans l’ADN (Methylation specific-MLPA (MS-MLPA)).
Limitations de la MLPA
- La MLPA est extrêmement sensible aux impuretés. Par conséquent, il faut être extrêmement prudent lors de la préparation des échantillons et de la réalisation de la technique.
- Il peut y avoir une diminution du signal d’une sonde en raison d’un polymorphisme ou d’une mutation rare, et il pourrait être nécessaire de le tester par d’autres techniques.
La MLPA est une excellente technique qui peut être utilisée pour des applications variées et qui donne des résultats rapidement et efficacement. Cependant, elle n’est pas sans accrocs. Comme nous le savons tous, chaque technique a certains inconvénients, et chaque application doit être étudiée en profondeur, pour s’assurer que nous utilisons la technique la plus efficace et la plus sensible.
Avez-vous déjà utilisé la MLPA ? Que pensez-vous de cette technique ?