La amplificación de sonda dependiente de ligadura múltiple (MLPA) es una técnica molecular desarrollada por el MRC-Holland en 2002. En pocas palabras, MLPA es una técnica sensible que permite la cuantificación de secuencias de ácidos nucleicos, de forma rápida y eficiente. Se realiza en muchos laboratorios de todo el mundo y puede aplicarse para detectar cambios en el número de copias (como deleciones o duplicaciones) de un gen, identificar el estado de metilación del ADN, detectar polimorfismos de un solo nucleótido (SNP) y mutaciones puntuales, y cuantificar el ARNm. Por lo tanto, se utiliza en muchos campos de investigación y diagnóstico, como la citogenética, la investigación del cáncer y la genética humana, entre otros.
¿Cómo funciona?
El MLPA consta de los siguientes pasos (Figura 1):
- Denaturalización
- Hibridación
- Ligación
- Amplificación (por PCR)
- Separación de fragmentos y análisis de datos
Figura 1 – Visualización de la técnica MLPA (adaptada de Schouten, Jan P., et al.1). También podemos observar un electroferograma típico obtenido mediante el análisis MLPA que muestra una deleción del exón 46 (flecha roja). (Electroferograma adaptado de https://commons.wikimedia.org/wiki/File:MLPA_in_GeneMarker.jpg)
1-Denaturalización y 2 – Hibridación
La desnaturalización implica la separación de las hebras de ADN recocidas, de modo que el ADN de doble cadena se convierte en monocatenario.
La hibridación implica la hibridación de la muestra de ADN con sondas específicas. Debido a que es una técnica multiplex, se puede analizar cada muestra con hasta 60 sondas simultáneamente, dirigiéndose así a diferentes sitios.
Estas sondas tienen una secuencia de cebador que se une al cebador de la PCR en el proceso de amplificación. Todas las sondas diferentes tendrán la misma secuencia de unión al cebador. Además, las sondas también tienen una secuencia de hibridación complementaria al sitio objetivo que permitirá que la sonda se una al ADN. Ambas sondas se hibridarán en sitios adyacentes de la cadena de ADN.
Una de las sondas del par contiene una secuencia de relleno, cuya longitud es diferente para cada sitio objetivo. La longitud de la secuencia de relleno cambia entre las diferentes sondas, lo que permite el multiplexado. Por lo tanto, se puede esperar que cada producto de amplificación tenga una longitud única
3-Ligación
El paso de ligación unirá las dos sondas. En este paso se utiliza una enzima específica llamada ADN ligasa. Ésta une las sondas que ya están hibridadas en sitios adyacentes de la cadena de ADN en el sitio objetivo. La ligasa utilizada en los protocolos MLPA es la ligasa-65, una enzima ligasa dependiente de NAD, que también puede ser útil en otras aplicaciones.
Ahora, la pregunta es: si nuestro objetivo es ligar ambas sondas, ¿por qué son moléculas separadas para empezar? Pues bien, ambas sondas contienen los sitios de unión para los cebadores de PCR. Esto significa que, si utilizáramos las sondas como una sola molécula, obtendríamos un producto de amplificación, incluso sin el sitio diana del ADN, lo que nos daría una amplificación no específica. La enzima ligasa es extremadamente específica: si hay algún desajuste entre la sonda y el sitio diana, la ligasa no podrá unir las sondas y no se produciría la amplificación. En consecuencia, el MLPA detecta mutaciones puntuales específicas, e incluso distingue entre pseudogenes y el verdadero gen diana.
4-Amplificación
El siguiente paso es la amplificación, que es esencialmente una reacción en cadena de la polimerasa (PCR) (Tabla 1). Para el paso de la PCR, se añade una polimerasa, dNTPs y un cebador directo e inverso. Dado que todas las sondas tienen la misma secuencia de cebador PCR, sólo será necesario añadir un par de cebadores universales para estudiar todas nuestras dianas. El cebador directo está marcado con fluorescencia, lo que permite su visualización y cuantificación durante el análisis.
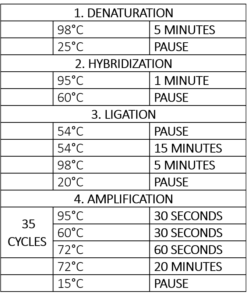
Tabla 1 – Programa del termociclador para la reacción MLPA
5-Separación de los fragmentos y análisis de los datos
Tras la amplificación, los fragmentos se separan por electroforesis capilar. La electroforesis capilar separa los fragmentos en función de su longitud, y muestra los fragmentos de diferente longitud como patrones de picos, llamados electroferograma (Figura 1). Cada amplicón tiene un tamaño conocido diferente, debido a la secuencia de relleno en cada sonda específica, y por lo tanto cada amplicón puede ser cuantificado durante el análisis de datos.
Los datos obtenidos por electroforesis capilar serán la entrada para el análisis. MRC- Holland proporciona un software gratuito para el análisis de datos – Coffalyser.
Al comparar cada muestra con un conjunto de muestras de referencia, podemos obtener una proporción de sonda. Esta proporción de sondas nos informará de cuántos números de copias tiene un gen. Dado que la mayoría de los genes humanos son diploides, si la muestra presenta dos copias, el ratio será de 1,0; es decir, las sondas de la muestra han obtenido el mismo número de genes que la muestra de referencia.
Sin embargo, si el ratio es de 0,5, sólo había una copia del gen en el individuo, lo que probablemente significa una deleción heterocigótica del gen objetivo. Si, por el contrario, la proporción es de 1,5, hay, probablemente, una duplicación heterocigótica de un gen.
MRC-Holland ofrece muchos kits diferentes que pueden tener la solución para sus problemas. Sin embargo, si lo que quieres es encontrar algo un poco más oscuro, o estudiar algo que no está en ningún kit, puedes diseñar tus propias sondas. Le aconsejo que lea detenidamente el protocolo para el diseño de sondas sintéticas.
Ventajas del MLPA
- El MLPA es una técnica muy sensible, robusta y de alto rendimiento.
- Puede discernir entre mutaciones puntuales, así como duplicación/deleción de genes. Por lo tanto, tiene una gran ventaja sobre otras técnicas, como la secuenciación, que sólo pueden encontrar mutaciones puntuales. Además, a diferencia del FISH, el MLPA puede detectar pequeñas alteraciones genéticas.
- Los resultados están disponibles en 24 horas y, al tratarse de una reacción multiplex, permite una recopilación de información rápida y eficiente.
- Pequeñas alteraciones en el protocolo del MLPA pueden permitir una gran variedad de aplicaciones. Por ejemplo, añadiendo un paso de digestión adicional, el MLPA también puede utilizarse para detectar patrones de metilación en el ADN (Methylation specific-MLPA (MS-MLPA)).
Limitaciones del MLPA
- El MLPA es extremadamente sensible a las impurezas. Por lo tanto, es necesario extremar la precaución a la hora de preparar las muestras y realizar la técnica.
- Puede haber una disminución de la señal de una sonda debido a un polimorfismo o una mutación poco frecuentes, y podría ser necesario analizarla mediante otras técnicas.
MLPA es una gran técnica que puede utilizarse para aplicaciones variadas y ofrece resultados de forma rápida y eficaz. Sin embargo, no viene sin contratiempos. Como todos sabemos, cada técnica tiene ciertas desventajas, y cada aplicación necesita ser estudiada a fondo, para asegurarse de que estamos utilizando la técnica más eficiente y sensible.
¿Has utilizado alguna vez el MLPA? ¿Qué opina de esta técnica?