Multiplex ligation-dependent probe amplification (MLPA) ist eine molekulare Technik, die 2002 vom MRC-Holland entwickelt wurde. Kurz gesagt, ist MLPA eine empfindliche Technik, die eine schnelle und effiziente Quantifizierung von Nukleinsäuresequenzen ermöglicht. Sie wird in vielen Labors weltweit durchgeführt und kann zum Nachweis von Veränderungen der Kopienzahl eines Gens (wie Deletionen oder Duplikationen), zur Bestimmung des Methylierungsstatus der DNA, zum Nachweis von Einzelnukleotid-Polymorphismen (SNPs) und Punktmutationen sowie zur Quantifizierung von mRNA eingesetzt werden. Daher wird es in vielen Forschungs- und Diagnosebereichen eingesetzt, unter anderem in der Zytogenetik, der Krebsforschung und der Humangenetik.
Wie funktioniert es?
MLPA besteht aus den folgenden Schritten (Abbildung 1):
- Denaturierung
- Hybridisierung
- Ligation
- Amplifikation (durch PCR)
- Fragmenttrennung und Datenanalyse
Abbildung 1 – Visualisierung der MLPA-Technik (adaptiert von Schouten, Jan P., et al.1). Wir können auch ein typisches Elektropherogramm sehen, das durch MLPA-Analyse erhalten wurde und eine Deletion von Exon 46 (roter Pfeil) zeigt. (Elektropherogramm nach https://commons.wikimedia.org/wiki/File:MLPA_in_GeneMarker.jpg)
1-Denaturierung und 2-Hybridisierung
Die Denaturierung beinhaltet die Trennung der vernetzten DNA-Stränge, so dass die doppelsträngige DNA einzelsträngig wird.
Bei der Hybridisierung wird die DNA-Probe mit spezifischen Sonden hybridisiert. Da es sich um ein Multiplex-Verfahren handelt, können Sie jede Probe mit bis zu 60 Sonden gleichzeitig analysieren und so verschiedene Stellen anvisieren!
Diese Sonden haben eine Primer-Sequenz, die an den PCR-Primer im Amplifikationsprozess bindet. Alle verschiedenen Sonden haben die gleiche Primer-Bindungssequenz. Zusätzlich haben die Sonden eine Hybridisierungssequenz, die komplementär zur Zielstelle ist und die Bindung der Sonde an die DNA ermöglicht. Beide Sonden hybridisieren an benachbarten Stellen auf dem DNA-Strang.
Eine der Sonden des Paares enthält eine Stuffer-Sequenz, die für jede Zielstelle unterschiedlich lang ist. Die Länge der Stuffer-Sequenz ändert sich zwischen den verschiedenen Sonden, was das Multiplexing ermöglicht. Sie können also davon ausgehen, dass jedes Amplifikationsprodukt eine einzigartige Länge hat!
3-Ligation
Der Ligationsschritt verbindet die beiden Sonden miteinander. In diesem Schritt wird ein spezielles Enzym namens DNA-Ligase verwendet. Es bindet die Sonden, die bereits an benachbarten Stellen des DNA-Strangs an der Zielstelle hybridisiert sind. Bei der in MLPA-Protokollen verwendeten Ligase handelt es sich um Ligase-65, ein NAD-abhängiges Ligaseenzym, das auch in anderen Anwendungen nützlich sein kann.
Nun stellt sich die Frage: Wenn unser Ziel darin besteht, beide Sonden zu ligieren, warum sind sie dann zunächst getrennte Moleküle? Nun, beide Sonden enthalten die Bindungsstellen für PCR-Primer. Das bedeutet, dass wir, wenn wir die Sonden als ein einziges Molekül verwenden würden, ein Amplifikationsprodukt erhalten würden, auch ohne die DNA-Zielstelle, was zu einer unspezifischen Amplifikation führt. Das Enzym Ligase ist äußerst spezifisch: Wenn zwischen der Sonde und der Zielstelle eine Fehlpaarung vorliegt, kann die Ligase die Sonden nicht binden, und es würde keine Amplifikation stattfinden. Folglich weist MLPA spezifische Punktmutationen nach und unterscheidet sogar zwischen Pseudogenen und dem eigentlichen Zielgen.
4-Amplifikation
Der nächste Schritt ist die Amplifikation, die im Wesentlichen eine Polymerase-Kettenreaktion (PCR) ist (Tabelle 1). Für den PCR-Schritt werden eine Polymerase, dNTPs und ein Vorwärts- und Rückwärtsprimer hinzugefügt. Da alle Sonden die gleiche PCR-Primersequenz haben, muss nur ein Paar universeller Primer hinzugefügt werden, um alle unsere Ziele zu untersuchen. Der Vorwärtsprimer ist fluoreszierend markiert, was die Visualisierung und Quantifizierung während der Analyse ermöglicht.
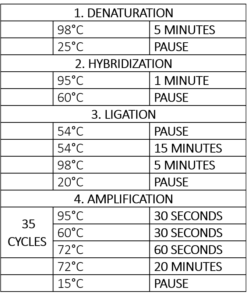
Tabelle 1 – Thermocycler-Programm für die MLPA-Reaktion
5-Fragmenttrennung und Datenanalyse
Nach der Amplifikation werden die Fragmente durch Kapillarelektrophorese getrennt. Die Kapillarelektrophorese trennt die Fragmente nach ihrer Länge und zeigt die Fragmente unterschiedlicher Länge als Peak-Muster, die als Elektropherogramm bezeichnet werden (Abbildung 1). Jedes Amplikon hat aufgrund der Stuffer-Sequenz auf jeder spezifischen Sonde eine andere bekannte Größe, so dass jedes Amplikon während der Datenanalyse quantifiziert werden kann.
Die durch Kapillarelektrophorese gewonnenen Daten dienen als Input für die Analyse. MRC-Holland stellt eine kostenlose Software für die Datenanalyse zur Verfügung – Coffalyser.
Durch den Vergleich jeder Probe mit einem Satz von Referenzproben können wir ein Sondenverhältnis erhalten. Dieses Sondenverhältnis gibt Aufschluss darüber, wie viele Kopien ein Gen hat. Da die meisten menschlichen Gene diploid sind, beträgt das Verhältnis 1,0, wenn die Probe zwei Kopien aufweist, d. h. die Probensonden haben die gleiche Anzahl von Genen erhalten wie die Referenzprobe.
Beträgt das Verhältnis jedoch 0,5, gab es nur eine Kopie des Gens in dem Individuum, was wahrscheinlich eine heterozygote Deletion des Zielgens bedeutet. Beträgt das Verhältnis hingegen 1,5, liegt wahrscheinlich eine heterozygote Duplikation eines Gens vor.
MRC-Holland bietet viele verschiedene Kits an, die möglicherweise die Lösung für Ihre Probleme bieten. Wenn Sie jedoch etwas suchen, das etwas undurchsichtiger ist, oder etwas untersuchen wollen, das in keinem Kit enthalten ist, können Sie Ihre eigenen Sonden entwerfen. Ich empfehle Ihnen, das Protokoll für das Design synthetischer Sonden sorgfältig zu lesen.
Vorteile von MLPA
- MLPA ist eine hochempfindliche, robuste Technik mit hohem Durchsatz.
- Sie kann sowohl Punktmutationen als auch Duplikationen/Deletionen von Genen unterscheiden. Daher hat sie einen großen Vorteil gegenüber anderen Techniken, wie der Sequenzierung, die nur Punktmutationen aufspüren können. Außerdem kann MLPA im Gegensatz zu FISH auch kleine Genveränderungen nachweisen.
- Die Ergebnisse liegen innerhalb von 24 Stunden vor, und da es sich um eine Multiplex-Reaktion handelt, können schnell und effizient Informationen gesammelt werden.
- Kleine Änderungen am MLPA-Protokoll können eine Vielzahl von Anwendungen ermöglichen. Durch Hinzufügen eines zusätzlichen Verdauungsschritts kann MLPA beispielsweise auch zum Nachweis von Methylierungsmustern in der DNA verwendet werden (Methylierungs-spezifische-MLPA (MS-MLPA)).
Grenzwerte von MLPA
- MLPA ist extrem empfindlich gegenüber Verunreinigungen. Daher ist bei der Vorbereitung der Proben und der Durchführung des Verfahrens äußerste Vorsicht geboten.
- Bei seltenen Polymorphismen oder Mutationen kann sich das Signal einer Sonde abschwächen, so dass es erforderlich sein kann, sie mit anderen Verfahren zu testen.
MLPA ist ein hervorragendes Verfahren, das für verschiedene Anwendungen eingesetzt werden kann und schnell und effizient Ergebnisse liefert. Allerdings ist sie nicht ohne Probleme. Wie wir alle wissen, hat jede Technik gewisse Nachteile, und jede Anwendung muss gründlich untersucht werden, um sicherzustellen, dass wir die effizienteste und empfindlichste Technik verwenden.
Haben Sie MLPA jemals verwendet? Was denken Sie über diese Technik?