Multiplex ligation-dependent probe amplification (MLPA) è una tecnica molecolare sviluppata da MRC-Holland nel 2002. In poche parole, la MLPA è una tecnica sensibile che permette di quantificare le sequenze di acidi nucleici, in modo rapido ed efficiente. Viene eseguita in molti laboratori in tutto il mondo e può essere applicata per rilevare cambiamenti nel numero di copie (come delezioni o duplicazioni) di un gene, identificare lo stato di metilazione del DNA, rilevare polimorfismi a singolo nucleotide (SNPs) e mutazioni puntiformi, e quantificare l’mRNA. Pertanto, è utilizzato in molti campi di ricerca e diagnostici, come la citogenetica, la ricerca sul cancro e la genetica umana, tra gli altri.
Come funziona?
MLPA consiste nelle seguenti fasi (Figura 1):
- Denaturazione
- Ibridizzazione
- Ligazione
- Amplificazione (tramite PCR)
- Separazione dei frammenti e analisi dei dati
Figura 1 – Visualizzazione della tecnica MLPA (adattata da Schouten, Jan P., et al.1). Possiamo anche osservare un tipico elettroferogramma ottenuto attraverso l’analisi MLPA che mostra una delezione dell’esone 46 (freccia rossa). (Elettroferogramma adattato da https://commons.wikimedia.org/wiki/File:MLPA_in_GeneMarker.jpg)
1-Denaturazione e 2 – Ibridazione
La denaturazione comporta la separazione dei filamenti di DNA ricotti, in modo che il DNA a doppio filamento diventi a filamento singolo.
L’ibridazione comporta l’ibridazione del campione di DNA a sonde specifiche. Poiché si tratta di una tecnica multiplex, è possibile analizzare ogni campione da un massimo di 60 sonde contemporaneamente, mirando così a siti diversi!
Queste sonde hanno una sequenza di primer che si lega al primer della PCR nel processo di amplificazione. Tutte le diverse sonde avranno la stessa sequenza di legame del primer. Inoltre, le sonde hanno anche una sequenza di ibridazione complementare al sito bersaglio che permetterà alla sonda di legarsi al DNA. Entrambe le sonde si ibrideranno su siti adiacenti sul filamento di DNA.
Una delle sonde della coppia contiene una sequenza stuffer, che è di lunghezza diversa per ogni sito bersaglio. La lunghezza della sequenza stuffer cambia tra le diverse sonde, permettendo il multiplexing. Quindi, ci si può aspettare che ogni prodotto di amplificazione abbia una lunghezza unica!
3-Ligazione
La fase di legatura legherà insieme le due sonde. In questa fase, viene utilizzato un enzima specifico chiamato DNA ligasi. Essa lega le sonde che sono già ibridate su siti adiacenti del filamento di DNA nel sito bersaglio. La ligasi usata nei protocolli MLPA è la ligasi-65, un enzima ligasi NAD-dipendente, che può essere utile anche in altre applicazioni.
Ora, la domanda è: se il nostro obiettivo è quello di legare entrambe le sonde, perché sono molecole separate per cominciare? Bene, entrambe le sonde contengono i siti di legame per i PCR-primer. Questo significa che se usassimo le sonde come una singola molecola, otterremmo un prodotto di amplificazione, anche senza il sito bersaglio del DNA, dandoci così un’amplificazione non specifica. L’enzima ligasi è estremamente specifico: se ci sono dei mismatch tra la sonda e il sito bersaglio, la ligasi non sarà in grado di legare le sonde e non avverrà alcuna amplificazione. Di conseguenza, la MLPA rileva mutazioni puntuali specifiche e distingue anche tra pseudogeni e il vero gene bersaglio.
4-Amplificazione
La fase successiva è l’amplificazione, che è essenzialmente una reazione a catena della polimerasi (PCR) (Tabella 1). Per la fase di PCR, vengono aggiunti una polimerasi, dNTP e un primer forward e reverse. Poiché tutte le sonde hanno la stessa sequenza PCR-primer, sarà necessario aggiungere solo una coppia di primer universali per studiare tutti i nostri obiettivi. Il primer forward è marcato con fluorescenza, permettendo la visualizzazione e la quantificazione durante l’analisi.
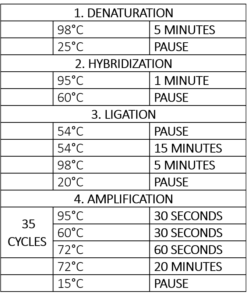
Tabella 1 – Programma del termociclatore per la reazione MLPA
5-Separazione dei frammenti e analisi dei dati
Dopo l’amplificazione, i frammenti sono separati per elettroforesi capillare. L’elettroforesi capillare separa i frammenti in base alla loro lunghezza e mostra i frammenti di lunghezza diversa come modelli di picchi, chiamati elettroferogramma (Figura 1). Ogni amplicone ha una dimensione diversa nota, a causa della sequenza stuffer su ogni sonda specifica, e quindi ogni amplicone può essere quantificato durante l’analisi dei dati.
I dati ottenuti dall’elettroforesi capillare saranno l’input per l’analisi. MRC-Olanda fornisce un software gratuito per l’analisi dei dati – Coffalyser.
Confrontando ogni campione con un set di campioni di riferimento, possiamo ottenere un rapporto di sonda. Questo rapporto di sonda ci informerà di quanti numeri di copie ha un gene. Poiché la maggior parte dei geni umani sono diploidi, se il campione presenta due copie, il rapporto sarà 1,0; cioè le sonde del campione hanno ottenuto lo stesso numero di geni del campione di riferimento.
Tuttavia, se il rapporto è 0,5 c’era solo una copia del gene nell’individuo, il che significa probabilmente una delezione eterozigote del gene target. Se, invece, il rapporto è 1,5, c’è, probabilmente, una duplicazione eterozigote di un gene.
MRC-Holland offre molti kit diversi che possono avere la soluzione ai vostri problemi. Tuttavia, se state cercando di trovare qualcosa di un po’ più oscuro, o di studiare qualcosa che non si trova in nessun kit, potete progettare le vostre sonde. Ti consiglio di leggere attentamente il protocollo per la progettazione di sonde sintetiche.
Svantaggi di MLPA
- MLPA è una tecnica altamente sensibile, robusta e ad alta produttività.
- Può discernere tra mutazioni puntiformi, così come la duplicazione/cancellazione di geni. Pertanto, ha un grande vantaggio rispetto ad altre tecniche, come il sequenziamento, che possono trovare solo mutazioni puntiformi. Inoltre, a differenza del FISH, l’MLPA può rilevare piccole alterazioni geniche.
- I risultati sono disponibili entro 24 ore e poiché si tratta di una reazione multiplex, consente una raccolta rapida ed efficiente di informazioni.
- Piccole alterazioni al protocollo MLPA possono consentire una varietà di applicazioni. Per esempio, aggiungendo un ulteriore passaggio di digestione, l’MLPA può anche essere usato per rilevare i modelli di metilazione nel DNA (Methylation specific-MLPA (MS-MLPA)).
Limitazioni dell’MLPA
- MLPA è estremamente sensibile alle impurità. Pertanto, è necessaria un’estrema cautela nella preparazione dei campioni e nell’esecuzione della tecnica.
- Può verificarsi una diminuzione del segnale di una sonda a causa di un polimorfismo o di una mutazione rara, e potrebbe essere necessario testarla con altre tecniche.
MLPA è una grande tecnica che può essere utilizzata per varie applicazioni e dà risultati in modo rapido ed efficiente. Tuttavia, non è priva di intoppi. Come tutti sappiamo, ogni tecnica ha alcuni svantaggi, e ogni applicazione deve essere studiata a fondo, per essere sicuri di usare la tecnica più efficiente e sensibile.
Hai mai usato MLPA? Quali sono i tuoi pensieri su questa tecnica?